最近、個人向けの3Dプリンタを買いました。折角なので自分の脳みそをプリントしてみようと思いました。
CT画像から骨を抽出してSTL出力するのはInVesaliusとかでちょちょっとやったら出来るけど、MRIからの皮質抽出は単純じゃないのでまとめてみた。
個人的な研究の為に使用してください。もしどこかから怒られたらこの記事は削除します。

目次
手順の概要
- MRI画像(DICOMデータ)を用意する
- MRIcroGLでDICOM→NIFTI形式に変換
- BrainSuiteで表面抽出
- 抽出したデータをWavefront OBJ形式で出力
- Fusion 360でSTL形式に変換
MRI画像(DICOMデータ)を用意する
MRI画像は自分のものを使用してください。
T1強調画像である事が必要です。可能な限り高解像度な方が望ましいですが、MPRAGEでなくても多分大丈夫です。
ファイルがないけど試してみたい!って人は、標準脳データを使ってみるのも良いかもしれません。通常のT1画像とは異なりますが、手元で試した感じうまく抽出できるようです。
手っ取り早く脳を印刷したい!という方は、Thingiverseのモデルを使うか、NIH 3D Print Exchangeで”full brain”を検索してみてください。
MRIcroGLでDICOM→NIFTI形式に変換
MRIcroGLは軽量の医用画像ビューアです。後述のBrainSuiteではDICOM形式を直接読み込むことが出来ないので、これを使ってDICOM形式(.dcm)をNIFTI形式(.nii/.nii.gz)に変換します。
MRIcroGLのダウンロード
以下のサイトからダウンロードします。インストール不要。
https://www.nitrc.org/frs/?group_id=889
圧縮ファイルを展開したら、MRIcroGL.exeを実行します。
DICOM→NIFTIへ変換
“Import”→”Convert DICOM to NIFTI”を選択し、設定はデフォルトのままで、出てきたウィンドウ上にDICOMファイルが入ったフォルダをドラッグ&ドロップします。
数分~数秒で変換が完了し、DICOMフォルダ内にnii.gzファイルとjsonファイルが作成されます。jsonファイルの方は使わないので削除してしまって構いません。
尚、ここで”File”→”Open Standard”→”mni152″を開いて”Save NIfTI”でエクスポートすると、標準脳を使った3Dデータを作成する事ができます。
BrainSuiteで表面抽出
BrainSuiteはFreeSurferのようにMRIデータからsurface dataなどを計算するオープンソースソフトウェアです。拡散テンソル画像なども扱えます。あくまで研究用のソフトウェアですので、必ず利用規約を読んでから使用してください。
今回これを選んだのは、ワンクリックで全ての処理を行えることと、デフォルトの設定が工夫されていて一定水準以上のクオリティを発揮できる事が多いためです。
BrainSuiteのインストール
以下のサイトからダウンロードします。インストール作業が必要です。
http://brainsuite.org/quickstart/installation/
尚、実行にはMATLAB Runtime(無料)のインストールも必要です。上記ページ内でダウンロード先も案内されているので、こちらを先にインストールしておきましょう。
NIFTIファイルの読み込みと表面抽出処理
BrainSuiteを起動したら、”Open a 3D image file”を選択し、先ほど変換したNIFTIファイル(.nii.gz)を読み込みます。
次に、”Cortex”→”Cortical Surface Extraction Sequence”を選択し、小ウィンドウを立ち上げます。
小ウィンドウ下部の”File name prefix”と”Working Directory”で、ファイルの保存先を指定します。数十ファイル作成されるので、専用のフォルダを作成した方が良いと思います。
他の設定は全部デフォルトのまま(左側のチェックボックスは”Register and label brain”以外すべてチェック)にして、小ウィンドウ右上の[Run All]を押すと表面抽出処理が開始されます。
コンピュータのスペックにもよりますが、処理には数十分~数時間かかります。
処理が完了したら、一度BrainSuiteを終了させてしまいましょう。
抽出したデータをWavefront OBJ形式で出力
保存先に指定したフォルダ内のファイルから、”[prefix].pial.cortex.dfs”を探します。[prefix]は自分が設定したプレフィックスが入ります。
再度BrainSuiteを起動し、このファイルをドラッグ&ドロップして読み込ませます。
表面抽出がうまくいっていれば、下記のように脳の表面が表示されるはずです。
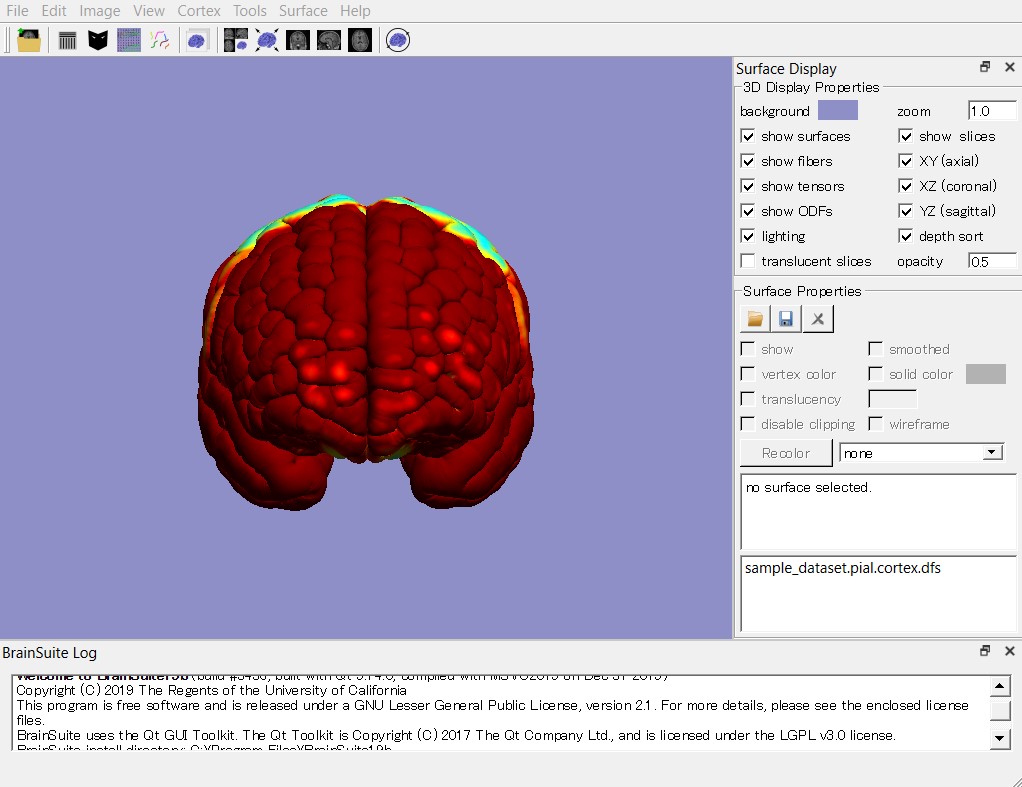
右側のツールボックス下部に、”[prefix].pial.cortex.dfs”が表示されているので、これをクリックして選択します。
次に、”Surface Properties”の下にある青い保存ボタンを押して保存ダイアログを表示させます。
保存先を選んだあと任意のファイル名を付けるのですが、このとき拡張子を”.wfo”として保存してください。”.obj”で保存すると、3Dモデルでよく使うWavefront OBJ形式ではなくMINI OBJ形式で保存されるようです。
保存したあと、拡張子を手動で”.wfo”から”.obj”に書き換えます。
出力したOBJファイル以外は削除してしまって構いませんが、「右半球だけ」「灰白質だけ」などのサーフェスデータもあるので、興味があれば使ってみてください。
Fusion 360でSTL形式に変換
私はAutodesk Fusion 360の個人版を使っていますが、ここからは好みのソフトウェアを使って結構です。
作成したOBJファイルを読み込み、必要に応じて拡大縮小やメッシュ数の削減などの加工を行って、STL形式でエクスポートしてください。
あとは、好みのスライサーで刻んで出力するだけです。
当然ですが、底面が水平ではないのでサポートが要ると思います。
出来そうだけどやってないこと
部位ごとに分割して出力したらパズルみたいにならないかな…と思ったけど、うまく輪切りしないと組み合わせられなくなりそうですね。
InVesaliusでやる方法も教えてください!
InVesaliusはマニュアルがしっかりしているので、こちらをご覧ください
https://invesalius.github.io/docs/user_guide/user_guide.html#segmentation